分子结构预测是生物信息学的“圣杯”,2024年诺贝尔化学奖授予了AI蛋白质结构预测系统AlphaFold的两位开发者,标志着人工智能(AI)预测方法能给生命科学研究带来革命性推进。然而,AlphaFold仍然未解决核酸结构预测的问题,RNA三维结构预测领域仍亟需明确其瓶颈和发展方向。
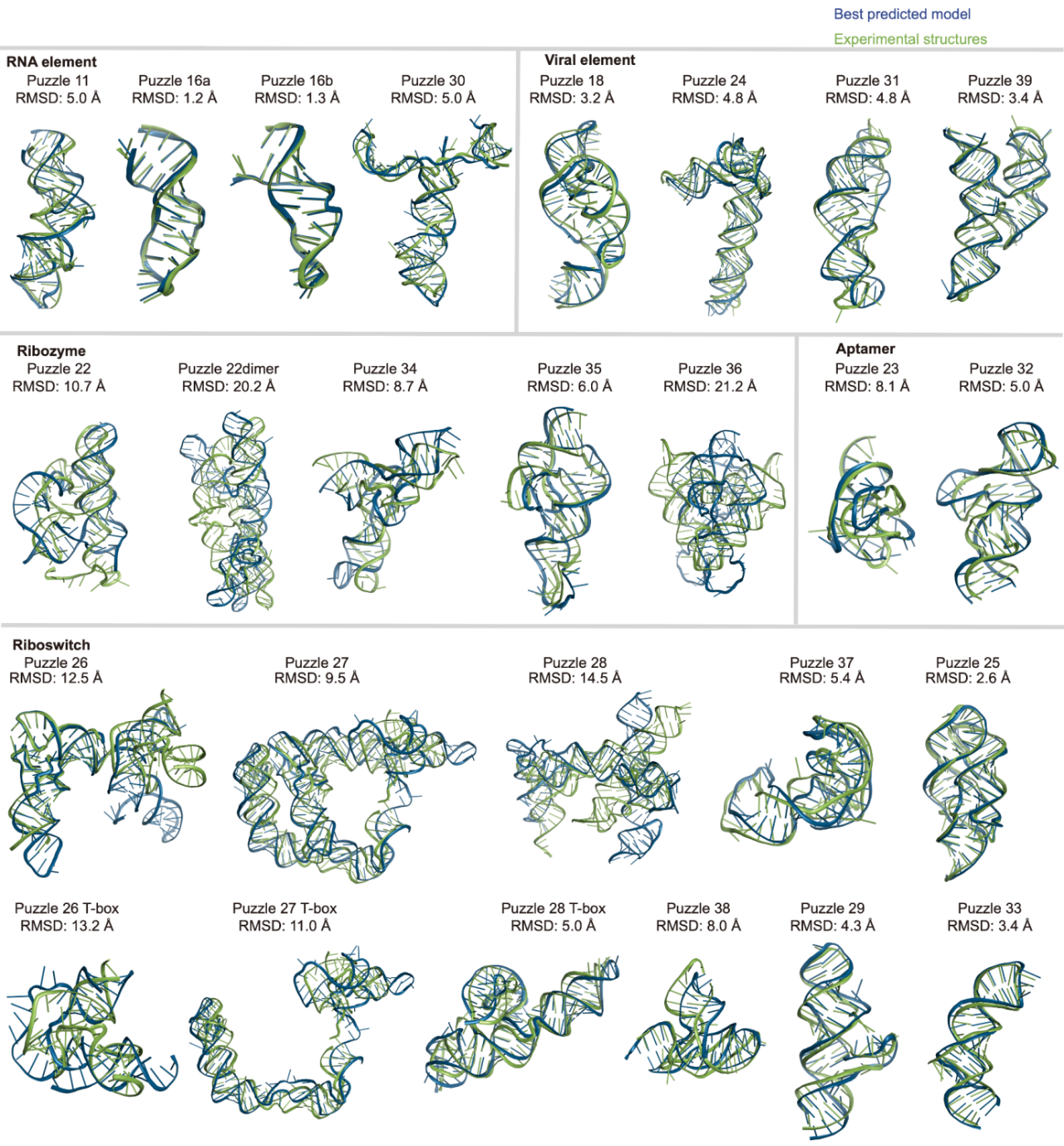
RNA-Puzzles是一个2012年开始的国际合作项目,致力于评估RNA三维结构预测的最新进展。组织者在RNA三维结构数据发表之前将需要预测的序列发给全球的结构预测团队,在预测结束后各个团队反馈预测结果,等三维结构数据发表以后将预测结构与实验测定的结构进行比较。从而评估RNA三维结构预测的准确性。 2024年12月2日,广州国家实验室、广医-广州生物院联合生科院苗智超教授团队联合中国科学院温州医学研究所 Eric Westhof 院士,在 Nature Methods 期刊发表了题为:RNA-Puzzles Round V:Blind predictions of 23 RNA structures 的论文,公布了RNA-Puzzles Round V 的结果,对来自全球18个团队的预测进行了大规模评估,涉及23个RNA结构,包括RNA元件、适配体、病毒元件、核酶和核开关等多种RNA类型。

本轮比赛(Round V)的预测结果显示,在RNA结构建模方面,一些关键步骤仍需克服。特别是,碱基配对的识别:准确识别形成RNA螺旋的碱基对仍然是一个挑战;non-Watson-Crick结构模块的识别:正确识别non-Watson-Crick碱基对和RNA结构模块对于精确建模至关重要;螺旋间的同轴堆积(coaxial):避免螺旋间的打结,并实现正确的同轴堆积是提高预测准确性的关键。论文还讨论了RNA结构预测的难度取决于是否存在同源模板或者预测序列的长度,链数等因素。
为了对预测结果进行客观评估,本轮比赛采用了多种评估指标,包括:均方根偏差(RMSD):用于衡量预测结构与实验结构之间的整体相似性,较低的RMSD值表示预测精度较高;相互作用网络保真度(INF):评估预测结构中碱基配对和碱基堆积相互作用的准确性;变形指数(DI):综合考虑RMSD和INF值,更全面地反映预测结构的质量;IDDT分数和ARES分数:分别侧重于局部和全局精度,以及RNA样结构特性的评估。这些算法工具和测评数据都已经开源,发布在RNA-Puzzles官网(www.rnapuzzles.org)。
本轮比赛中,排名前四的团队中有三个团队也曾在CASP15蛋白质结构预测比赛的RNA赛道中名列前茅,这表明前几名的预测方法具有较好的稳定性。尤其是陈世杰教授和Rhiju Das团队,陈世杰教授也在CASP16的比赛中获得全球第一,但不同预测方法在具体表现上差异不大。未来RNA结构预测领域有望借助人工智能结合经典物理模拟方法和专家经验实现进一步突破。
论文详细分析了不同功能类型RNA的预测结果,具体有——RNA元件:对一些简单的RNA元件,预测结果较为理想;适配体:对与配体结合的适配体,预测精度相对较低,尤其是在识别配体结合位点方面;病毒元件:一些病毒RNA元件的预测结果较好,但也存在一些预测精度较低的情况,尤其是一些包含假结的结构;核酶:对核酶的催化位点残基的预测,准确性与RMSD值密切相关;核开关:对一些具有同源结构的核开关,预测结果较好;而对于一些复杂的核开关,例如T-box核开关,预测精度仍然有待提高。
论文链接:https://www.nature.com/articles/s41592-024-02543-9



合作咨询
![]() 肖女士
肖女士
![]() 021-33392297
021-33392297
![]() Kelly.Xiao@imsinoexpo.com
Kelly.Xiao@imsinoexpo.com
 2006-2025 上海博华国际展览有限公司版权所有(保留一切权利)
沪ICP备05034851号-57
2006-2025 上海博华国际展览有限公司版权所有(保留一切权利)
沪ICP备05034851号-57